Une équipe de la filiale de Google DeepMind affirme que son réseau d’intelligence artificielle (IA) a fait un énorme pas en avant dans la résolution de Problème de repliement des protéines vieux de 50 ans après avoir battu toutes les autres équipes lors d’un défi de prédiction de la structure des protéines. Le programme a été accueilli avec enthousiasme par des chercheurs du monde entier qui disent qu’il pourrait révolutionner la biologie, dans des domaines particuliers tels que la conception de médicaments ou la durabilité environnementale.
Percée scientifique fantastique. L’un des plus gros problèmes de la science (repliement des protéines) semble avoir été résolu par l’intelligence artificielle. D’énormes implications pour la médecine. Toutes nos félicitations @DeepMind #AlphaFold https://t.co/qwbiJINMBh
– Jim Al-Khalili (@jimalkhalili) 30 novembre 2020
L’algorithme de Google, appelé AlphaFold2, a été le vainqueur incontesté de cette année Évaluation critique de la prévision de structure (Casp14) challenge – un concours biennal créé pour évaluer les progrès dans la précision de la prédiction de la structure des protéines par ordinateur AlphaFold2 a pu déterminer les formes 3D d’environ deux tiers des protéines cibles avec une précision comparable à celle des expériences en laboratoire, surpassant largement une centaine d’autres équipes.
Les protéines sont des molécules complexes composées de chaînes d’acides aminés qui peuvent se plier en différentes formes. Prédire à quoi ressemblera la structure 3D finale est un défi sur lequel les scientifiques travaillent depuis des décennies. Mohammed AlQuraishi de l’Université de Columbia, États-Unis, qui a développé l’un des algorithmes concurrents, pense que l’IA de DeepMind pourrait aider les chercheurs à comprendre les fonctions des protéines en comparant leurs structures prédites à des protéines dont la fonction et la forme sont connues. Il ajoute que bien que le programme n’ait pas été développé pour la conception de protéines, l’expérience d’autres modèles comme Rosetta suggère qu’il serait peut-être possible de l’adapter à cette fin. «Ce n’est probablement pas encore assez précis pour les applications médicales axées sur les petites molécules, mais cela peut aider à concevoir des anticorps pour les thérapies à base de protéines», dit-il.
Andrei Lupas à l’Institut Max Planck pour la biologie du développement en Allemagne, qui était l’un des juges de Casp14, estime qu’il y a beaucoup de magie algorithmique dans les coulisses. «Ils n’ont pas été trop clairs sur ce qu’ils font, mais ils passent beaucoup de temps à obtenir les bons détails, et cela semble constituer une très, très bonne prédiction globale», note-t-il. «Mon département a fourni un objectif pour une protéine que nous n’avions pas été en mesure de résoudre depuis une décennie. Ils nous ont donné un modèle avec lequel nous avons résolu la structure en une demi-heure!
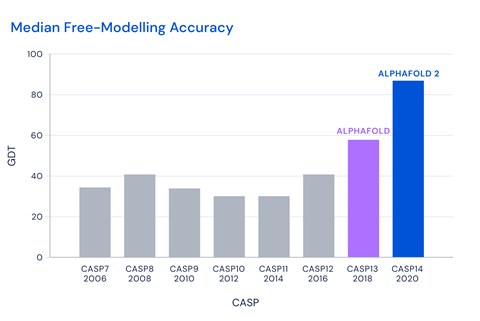
Le concours Casp utilise la métrique du test de distance globale (GDT) pour évaluer la précision. Tout programme atteignant un score d’environ 90 GDT est considéré comme compétitif avec les méthodes expérimentales. AlphaFold2 a atteint un score médian de 92,4 GDT sur toutes les cibles, laissant tous ses concurrents loin derrière. Le logiciel, qui a été formé sur environ 170000 structures du banque de données sur les protéines, s’appuie sur une version précédente présentée au Casp13 en 2018.
Il y a une différence essentielle entre les deux versions, explique AlQuraishi. «Celui-ci est différentiable de bout en bout, ce qui signifie que le système est optimisé pour passer de la séquence à la structure 3D finale, et que toutes les pièces du système sont optimisées conjointement pour apprendre des données», dit-il. «L’AlphaFold original avait plusieurs pièces séparées entraînées indépendamment et ne prédit que les distances interatomiques – pas les structures 3D – qui ont été utilisées pour plier la protéine en utilisant des approches plus conventionnelles comme Rosetta. AlphaFold2 est itératif, générant une structure 3D initiale qui est ensuite affinée en plusieurs étapes, «afin qu’il soit capable d’extraire des modèles plus complexes à partir des données», explique-t-il.
Bien que le modèle d’AlQuraishi n’ait pas fonctionné aussi bien qu’AlphaFold2, il s’est également amélioré par rapport à la version précédente. «Notre méthode fonctionne à partir de séquences de protéines individuelles, et non à partir de séquences de protéines homologues comme AlphaFold2. Nous pensons que cette voie en vaut la peine car elle peut permettre la conception de protéines très différentes de celles naturelles et peut être plus sensible aux changements dans les séquences individuelles, par exemple les mutations.
DeepMind souhaite maintenant améliorer davantage l’algorithme pour lui faire comprendre comment les protéines forment des complexes ou comment elles interagissent avec de petites molécules.








